摘要
大语言模型(LLMs)在通用医学(general medical)基准测试中表现优异,但其针对罕见病的推理能力尚未明确。
本研究并未采用 “让大语言模型诊断少量难以代表所有罕见病或罕见病相关基因的病例” 这一方式,而是致力于全面探究大语言模型对罕见病相关基因及表型的认知程度。
研究人员系统性评估了 6 款主流通用领域大语言模型(GPT-4、Claude 3.7、Llama-3.3 70B、Gemma-2 27B、Llama-3.2、Phi-4),测试其针对 Orphanet 收录的 10892 种罕见病,生成支撑疾病推理所需的核心表型特征及致病基因的能力。
研究将模型输出结果映射至 HPO 术语及人类基因命名委员会(HGNC)基因符号,并通过集合重叠度、语义相似度分析,以及基于 LIRICAL 分析框架对 8000 份患者表型数据包开展的疾病排序验证,与人工精选的参考数据进行对比。
结果显示,大语言模型对已整理罕见病知识的召回率整体偏低,且其对基因关联信息的检索准确性高于表型信息。商业模型(尤其是 GPT-4 与 Claude)的基因关联召回率超过 60%,但在精准还原表型信息方面表现欠佳。尽管模型输出与参考数据的精确重叠度较低,但中等水平的语义相似度得分表明二者存在部分一致性。
将大语言模型生成的表型特征谱应用于 LIRICAL 分析框架时,其疾病排序性能与金标准特征谱相近,不过直接诊断准确率仍有限。值得关注的是,不同模型输出的趋同性非整理术语,显示出大语言模型在罕见病假说生成方面具备潜在价值。
综上,当前通用大语言模型的精准度仍不足以替代人工整理的罕见病知识库,但可提供具有语义关联性的补充信息。本研究结果表明,融合专家人工整理数据与经筛选的大语言模型输出结果的混合策略,有助于优化并拓展基于本体论的罕见病诊断方法。
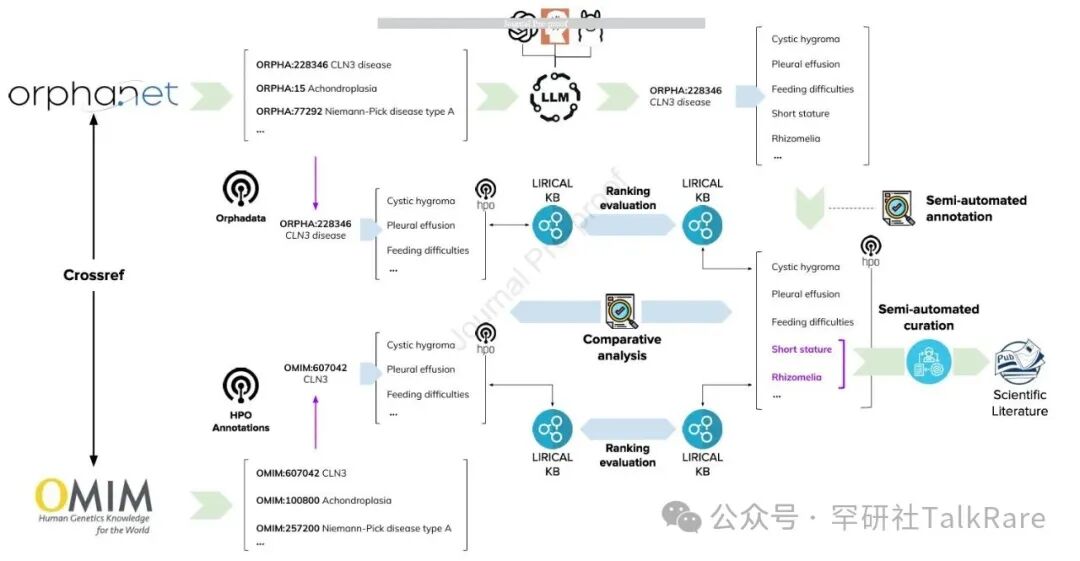 图 1 表型相关研究方法的整体概览
图 1 表型相关研究方法的整体概览
本研究选取 6 款大语言模型,以 Orphanet 收录的 10892 种疾病条目为研究对象,将疾病名称及对应同义术语(若有)作为输入提示,促使模型输出其存储的相关疾病知识。
研究人员将模型输出结果标准化映射至 HPO 术语集,并与源自 Orphanet 数据库及人类表型本体注释库(HPOA)的疾病 - 表型关联数据进行比对。同时,从 PubMed 摘要中提取相关证据,为新发现的疾病 - 表型关联关系提供支撑。
最后,将各模型生成的标注集作为知识库,依托 LIRICAL 分析框架内置的排序机制开展验证,并采用表型数据包的金标准语料库完成测试。
针对基因相关的研究流程与上述表型研究基本一致,差异点在于:其一,不执行 LIRICAL 排序分析;其二,采用人工方式,基于 HGNC 基因编号完成数据标准化处理。
